To pool or not to pool? (NL)
Doctoral fellow Jasper writes a blog for Eos Magazine
Om sneller en meer coronatesten te kunnen uitvoeren, is het mogelijk om stalen in groepen te testen in plaats van elk staal afzonderlijk. Een nadeel van die methode is dat meer resultaten valsnegatief zijn. Onderzoeker en blogger Jasper Verwilt (UGent) legt het dilemma uit.
Covid-19 is sinds maart voor veel mensen uitgegroeid tot een belangrijk onderdeel van hun dagelijks leven en heeft ondertussen meer dan 600 000 personen het leven gekost. Om de testcapaciteit te verhogen bekijkt men de optie stalen te groeperen (‘pooling’) voor het testen in plaats van de elk staal afzonderlijk te testen op het coronavirus. Dit is handig en snel, maar sommige stalen glippen zo door de mazen van het net. Anders gezegd: sommige mensen die corona hebben zullen dit niet te horen krijgen.
‘Pooling’ in de corona-toolbox
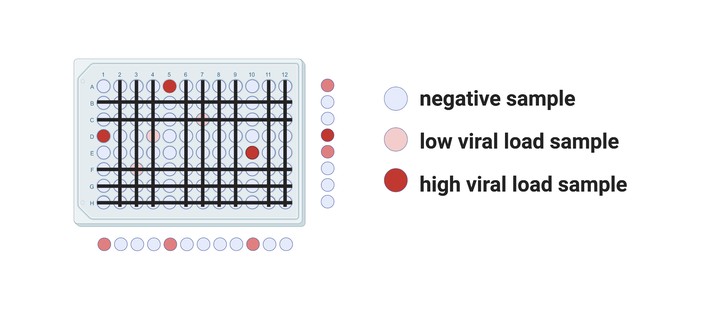
Om mensen te testen op het coronavirus wordt een neusswab genomen. Dit houdt in dat een lang waterstaafje enkele seconden in de neusholte gedraaid wordt (aangenaam is het niet), waarna de swab naar een testlabo wordt getransporteerd. In het labo wordt dan meestal een RT-qPCR-test uitgevoerd. Dit is een test die ons een idee geeft hoeveel virussen er aanwezig zijn in de swab. Sinds het begin van de covid-19 pandemie zijn er in België meer dan één miljoen van deze testen uitgevoerd. Dat is veel, maar als we de verspreiding van het coronavirus nog nauwkeuriger willen volgen, zal dit aantal nog zeer sterk moeten stijgen. Op dat moment duikt er echter een belangrijk probleem op: te veel testen, te weinig tijd, te weinig reagens. Om dit te vermijden zijn er labo’s in de VS en Israël overgestapt naar het zogenaamde ‘poolen’. Hierbij gaan we verschillende swabs samennemen — het aantal swabs per groep kan variëren—en testen we heel die groep in één keer. Het idee is dat wanneer in minstens één van de swabs virus aanwezig is, het virus ook aanwezig zal zijn in de groep, die vervolgens een positief resultaat zal geven. Enkel in groepen die positief testen, testen we elke staal individueel, terwijl we de stalen in de negatieve groepen niet opnieuw testen en als negatief labelen. Als we in België meer personen willen testen op covid-19, lijkt pooling op het eerste zicht een goede optie.
De twee strategieën die onderzoekers het meest naar voor schuiven zijn 1D en 2D pooling. Bij 1D pooling zit elk staal in één pool, bij 2D pooling zit elk staal in twee pools. Het verschil is het duidelijkst wanneer je de stalen organiseert in een matrix zoals weergegeven in de figuur.

Efficiëntie en sensitiviteit: een trade-off
Beide pooling strategieën zijn zeer krachtig en kunnen het aantal uit te voeren testen heel snel naar beneden halen: ‘pooling’ is efficiënter dan individueel testen. Als het allemaal zo simpel was, zou natuurlijk elk labo ter wereld hun swabs poolen. Dit is verre van de huidige situatie en de reden daarvoor zijn valsnegatieve stalen. Wanneer we een positieve swab met andere negatieve swabs poolen, verdunnen we het positieve staal. Dit kan zo sterk verdund worden dat we geen virus meer terugvinden in de pool, hoewel dit wel aanwezig was in het individueel staal. Heel de pool wordt verworpen en iedereen krijgt te horen dat hun test negatief was, ook die ene persoon met een positieve swab: een valsnegatieve. Dit gebeurt echter vooral als de stalen weinig virus bevatten. We rapporteren het aantal valsnegatieven meestal als ‘sensitiviteit’. Dit wordt berekend als het aantal swabs dat positief getest wordt, gedeeld door het werkelijk aantal positieve swabs. Een sensitiviteit van bijvoorbeeld 0,9 komt dus overeen met aanwezigheid van 10% valsnegatieven.
In de volgende figuur zien we een voorbeeld van hoe een efficiëntere strategie minder sensitief kan zijn. Er lijkt als het ware een soort trade-off (afweging) te zijn tussen beide parameters ofwel: ’t is ’t één of ‘t ander. Om na te gaan hoe deze trade-off zich juist gedraagt bij een variërend aantal positieve stalen en verschillende pooling strategieën, simuleerden we een hele hoop verschillende situaties met een computerprogramma.

Welke strategie is nu het beste?
We simuleerden het effect op sensitiviteit en efficiëntie van de pooling strategieën voor variërende percentages positieve stalen (prevalentie) van 0,01 tot 10%. De prevalentie kan verschillen naargelang de oorsprong van het staal. Ziekenhuizen hebben typisch een hoge prevalentie, terwijl de meeste testen uit woonzorgcentra negatief zijn en de prevalentie daar dus laag is. De RT-qPCR-test is heel gevoelig, dus we gaan er in de simulaties van uit dat als de stalen individueel getest worden, elk positief staal gevonden zal worden.
We zien dat als we elke Belg zouden willen testen (we kunnen dus een lage prevalentie verwachten), grote 1D pools (12, 16 of 24 stalen) het efficiëntst zijn. Het probleem is dat bij die lage prevalentie, de sensitiviteit van die poolingstrategieën problematisch laag is. Voor een 1D pooling met 24 stalen per groep bijvoorbeeld, zouden we één op vier positieve stalen negatief verklaren. Misschien is het dus beter van een beetje efficiëntie op te geven voor wat meer sensitiviteit en met pools van vier samples aan de slag gaan? Dit is de trade-off waar ik het eerder over had.
Hoe komt het eigenlijk dat de sensitiviteit van poolingstrategieën met grote pools heel erg afhangt van de prevalentie? Wanneer er heel weinig positieve stalen zijn en er zit één positief staal in een pool, is de kans namelijk klein dat er nog een tweede positief staal in de pool zal zitten. Het ene staal wordt verdund door alle negatieve stalen en raakt verloren in de analyse. Aan de andere kant, als er veel positieve stalen zijn, zit er bijna altijd meer dan één positief staal in een pool en test de pool dus meestal ook positief. De prevalentie is dus van groot belang, maar hoe berekenen we dat? Door de stalen te testen. Maar voor we testen moeten we weten wat de prevalentie is om de juiste strategie te achterhalen. Ziet u het probleem? Zo kunnen we cirkeltjes blijven draaien.
Wat nu?
Moeten we binnenkort covid-19 testen beginnen poolen? Zoals je wel hebt kunnen lezen is dat een lastige vraag en één die we jammer genoeg nog niet kunnen beantwoorden. Poolen is een heel krachtig trucje om snel veel stalen te analyseren, maar we zullen onvermijdelijk een paar positieve stalen over het hoofd zien (zeker bij lage prevalentie). Dit is trouwens zo met veel testen. Of we ons dat kunnen permitteren als we de volledige bevolking willen testen op corona, is een belangrijke afweging die ik gelukkig niet hoef te maken.
Meer lezen?
Onze resultaten hebben we online gezet in de vorm van een
preprint. Voordat die gepubliceerd wordt, moet die nog peer-review ondergaan. Tijdens de peer-review wordt het artikel grondig nagelezen en worden de auteurs van het artikel vaak gevraagd om aanpassingen te maken. Het is belangrijk om te weten dat dit hier dus nog niet gebeurd is. Een preprint dient altijd met een kritisch oog gelezen te worden.
Original post can be found
here.
After peer-review, this study was published in Scientific Reports and can be found
here.
‘Pooling’ in de corona-toolbox
Om mensen te testen op het coronavirus wordt een neusswab genomen. Dit houdt in dat een lang waterstaafje enkele seconden in de neusholte gedraaid wordt (aangenaam is het niet), waarna de swab naar een testlabo wordt getransporteerd. In het labo wordt dan meestal een RT-qPCR-test uitgevoerd. Dit is een test die ons een idee geeft hoeveel virussen er aanwezig zijn in de swab. Sinds het begin van de covid-19 pandemie zijn er in België meer dan één miljoen van deze testen uitgevoerd. Dat is veel, maar als we de verspreiding van het coronavirus nog nauwkeuriger willen volgen, zal dit aantal nog zeer sterk moeten stijgen. Op dat moment duikt er echter een belangrijk probleem op: te veel testen, te weinig tijd, te weinig reagens. Om dit te vermijden zijn er labo’s in de VS en Israël overgestapt naar het zogenaamde ‘poolen’. Hierbij gaan we verschillende swabs samennemen — het aantal swabs per groep kan variëren—en testen we heel die groep in één keer. Het idee is dat wanneer in minstens één van de swabs virus aanwezig is, het virus ook aanwezig zal zijn in de groep, die vervolgens een positief resultaat zal geven. Enkel in groepen die positief testen, testen we elke staal individueel, terwijl we de stalen in de negatieve groepen niet opnieuw testen en als negatief labelen. Als we in België meer personen willen testen op covid-19, lijkt pooling op het eerste zicht een goede optie.
De twee strategieën die onderzoekers het meest naar voor schuiven zijn 1D en 2D pooling. Bij 1D pooling zit elk staal in één pool, bij 2D pooling zit elk staal in twee pools. Het verschil is het duidelijkst wanneer je de stalen organiseert in een matrix zoals weergegeven in de figuur.

Efficiëntie en sensitiviteit: een trade-off
Beide pooling strategieën zijn zeer krachtig en kunnen het aantal uit te voeren testen heel snel naar beneden halen: ‘pooling’ is efficiënter dan individueel testen. Als het allemaal zo simpel was, zou natuurlijk elk labo ter wereld hun swabs poolen. Dit is verre van de huidige situatie en de reden daarvoor zijn valsnegatieve stalen. Wanneer we een positieve swab met andere negatieve swabs poolen, verdunnen we het positieve staal. Dit kan zo sterk verdund worden dat we geen virus meer terugvinden in de pool, hoewel dit wel aanwezig was in het individueel staal. Heel de pool wordt verworpen en iedereen krijgt te horen dat hun test negatief was, ook die ene persoon met een positieve swab: een valsnegatieve. Dit gebeurt echter vooral als de stalen weinig virus bevatten. We rapporteren het aantal valsnegatieven meestal als ‘sensitiviteit’. Dit wordt berekend als het aantal swabs dat positief getest wordt, gedeeld door het werkelijk aantal positieve swabs. Een sensitiviteit van bijvoorbeeld 0,9 komt dus overeen met aanwezigheid van 10% valsnegatieven.
In de volgende figuur zien we een voorbeeld van hoe een efficiëntere strategie minder sensitief kan zijn. Er lijkt als het ware een soort trade-off (afweging) te zijn tussen beide parameters ofwel: ’t is ’t één of ‘t ander. Om na te gaan hoe deze trade-off zich juist gedraagt bij een variërend aantal positieve stalen en verschillende pooling strategieën, simuleerden we een hele hoop verschillende situaties met een computerprogramma.

Welke strategie is nu het beste?
We simuleerden het effect op sensitiviteit en efficiëntie van de pooling strategieën voor variërende percentages positieve stalen (prevalentie) van 0,01 tot 10%. De prevalentie kan verschillen naargelang de oorsprong van het staal. Ziekenhuizen hebben typisch een hoge prevalentie, terwijl de meeste testen uit woonzorgcentra negatief zijn en de prevalentie daar dus laag is. De RT-qPCR-test is heel gevoelig, dus we gaan er in de simulaties van uit dat als de stalen individueel getest worden, elk positief staal gevonden zal worden.
We zien dat als we elke Belg zouden willen testen (we kunnen dus een lage prevalentie verwachten), grote 1D pools (12, 16 of 24 stalen) het efficiëntst zijn. Het probleem is dat bij die lage prevalentie, de sensitiviteit van die poolingstrategieën problematisch laag is. Voor een 1D pooling met 24 stalen per groep bijvoorbeeld, zouden we één op vier positieve stalen negatief verklaren. Misschien is het dus beter van een beetje efficiëntie op te geven voor wat meer sensitiviteit en met pools van vier samples aan de slag gaan? Dit is de trade-off waar ik het eerder over had.
Hoe komt het eigenlijk dat de sensitiviteit van poolingstrategieën met grote pools heel erg afhangt van de prevalentie? Wanneer er heel weinig positieve stalen zijn en er zit één positief staal in een pool, is de kans namelijk klein dat er nog een tweede positief staal in de pool zal zitten. Het ene staal wordt verdund door alle negatieve stalen en raakt verloren in de analyse. Aan de andere kant, als er veel positieve stalen zijn, zit er bijna altijd meer dan één positief staal in een pool en test de pool dus meestal ook positief. De prevalentie is dus van groot belang, maar hoe berekenen we dat? Door de stalen te testen. Maar voor we testen moeten we weten wat de prevalentie is om de juiste strategie te achterhalen. Ziet u het probleem? Zo kunnen we cirkeltjes blijven draaien.
Wat nu?
Moeten we binnenkort covid-19 testen beginnen poolen? Zoals je wel hebt kunnen lezen is dat een lastige vraag en één die we jammer genoeg nog niet kunnen beantwoorden. Poolen is een heel krachtig trucje om snel veel stalen te analyseren, maar we zullen onvermijdelijk een paar positieve stalen over het hoofd zien (zeker bij lage prevalentie). Dit is trouwens zo met veel testen. Of we ons dat kunnen permitteren als we de volledige bevolking willen testen op corona, is een belangrijke afweging die ik gelukkig niet hoef te maken.
Meer lezen?
Onze resultaten hebben we online gezet in de vorm van een preprint. Voordat die gepubliceerd wordt, moet die nog peer-review ondergaan. Tijdens de peer-review wordt het artikel grondig nagelezen en worden de auteurs van het artikel vaak gevraagd om aanpassingen te maken. Het is belangrijk om te weten dat dit hier dus nog niet gebeurd is. Een preprint dient altijd met een kritisch oog gelezen te worden.
Original post can be found here.
After peer-review, this study was published in Scientific Reports and can be found here.